はじめに
最近この本で勉強しています。
グラフを描画する際に、一緒に文字や直線、短径を描画したい時があると思います。
これらはデータそのものを描画しているわけではないのですが、グラフを解釈する際に有用になります。
今回はRでグラフないに文字や直線、短径を描画する方法を記載します。
目的
グラフ内に文字や直線、短径を描画する。
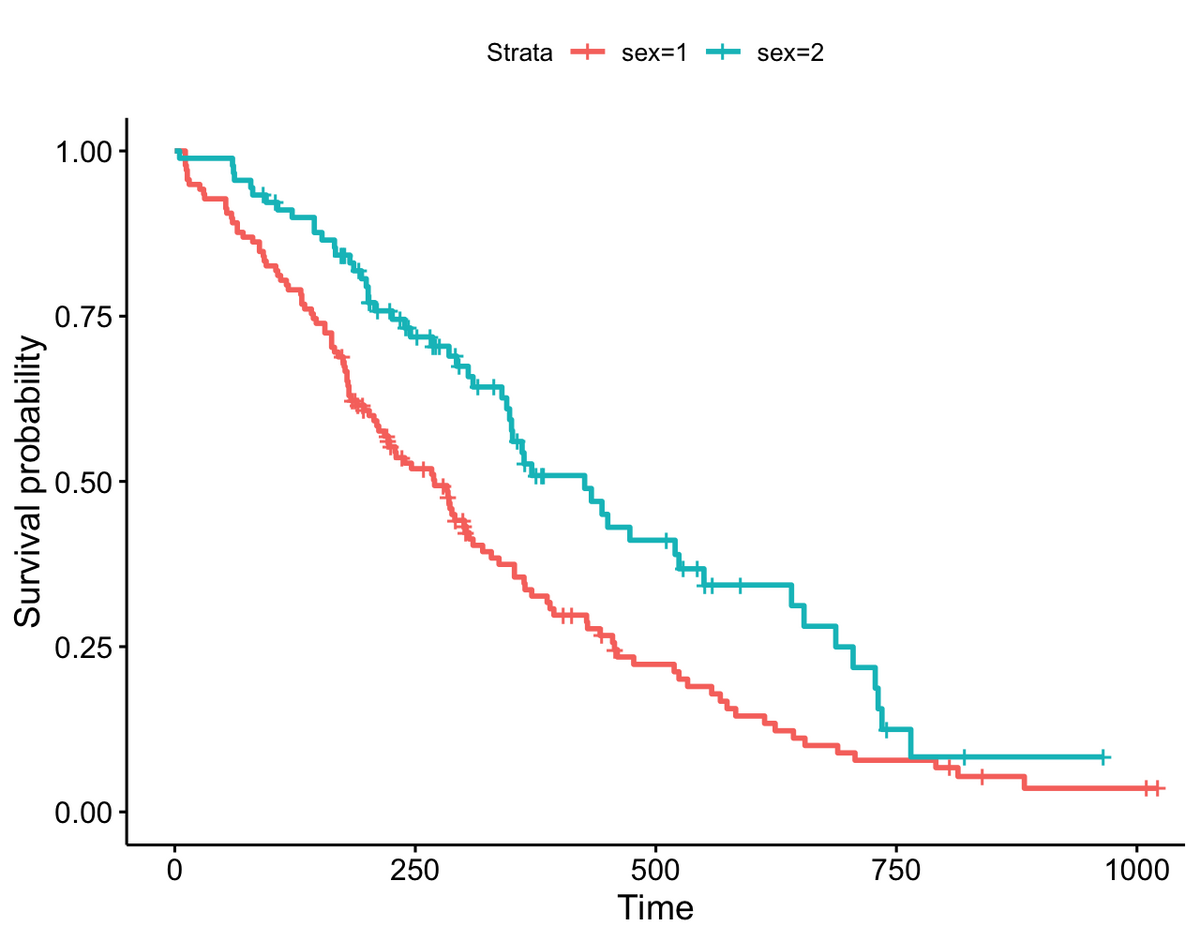
以下のようなグラフを作れるようになる。
使用するデータのチェック
お馴染みのiris (アヤメ) データを使用します。
データの中身を確認します。
irisデータセットはデフォルトで入っているので、一行で読み込めます。
library(tidyverse) iris

各行が1サンプルに対応していて、Sepal.Length, Sepal.Width, Petal.Length, Petal.Width, Speciesが与えられています。1
Species (アヤメの種類) 以外は数値データです。
ここでは、Petal.Length とPetal.Widthの関係に着目してみます。
この二つに正の相関があるのではないかという仮説をもとに可視化してみることにします。
データの前処理
最初に Spealのデータは今回使用しないので、selectで最初に除去します。
dat <- iris %>% select(-c(Sepal.Length, Sepal.Width))
シンプルなグラフの描画
まずは単純に描画してみます。
p <- ggplot(dat,
mapping = aes(x = Petal.Width,
y = Petal.Length,
color = Species))
p +
geom_point() +
theme_classic()

ThemeはClassicが好みです。 このグラフに、文字や直線、短径を追加してみます。
グラフ要素の追加
グラフ要素の追加はannotateを使って行います。
annotateの最初の引数には、追加する要素の種類を指定します。
- "test": 文字
- "segment": 線分
- "pointrange": 点と線分
- "rect": 四角形
文字の追加
最初は文字を追加してみます。
p +
geom_point() +
theme_classic() +
annotate("text", x = 0.5, y = 5, label = "Scatter plot", color = "black", size = 5)

x, y は文字の位置を指定します。文字の位置は軸の値で指定しています。
その他色やサイズも指定可能です。
線分の追加
適当な線分を追加する場合にはsegmentを用います。 この線分の使い道としては、グラフ上に閾値や平均値を示すといったところでしょうか。
p +
geom_point() +
annotate("segment", x = 0, xend = 3, y = 4, yend = 4, color = "gray", size = 1)+
annotate("segment", x = 1, xend = 1, y = 0, yend = 8, color = "gray", size = 1)+
theme_classic()

そのまま描画すると軸が浮いてしまって不格好なので、scale_x_continuous, scale_y_continuousでexpand = c(0, 0)を指定してきちんとくっつけます。
p +
geom_point() +
scale_x_continuous(expand = c(0, 0)) +
scale_y_continuous(expand = c(0, 0)) +
annotate("segment", x = 0, xend = 3, y = 4, yend = 4, color = "gray", size = 1)+
annotate("segment", x = 1, xend = 1, y = 0, yend = 8, color = "gray", size = 1)+
theme_classic()

もしくは今回のデータではあまり意味がありませんが、y=xの直線を引くことで縦軸と横軸の値がどちらが大きいのかということを示すのにも使えます。
p +
geom_point() +
scale_x_continuous(expand = c(0, 0)) +
scale_y_continuous(expand = c(0, 0)) +
annotate("segment", x = 0, xend = 3, y = 0, yend = 3, color = "gray", size = 1)+
theme_classic()

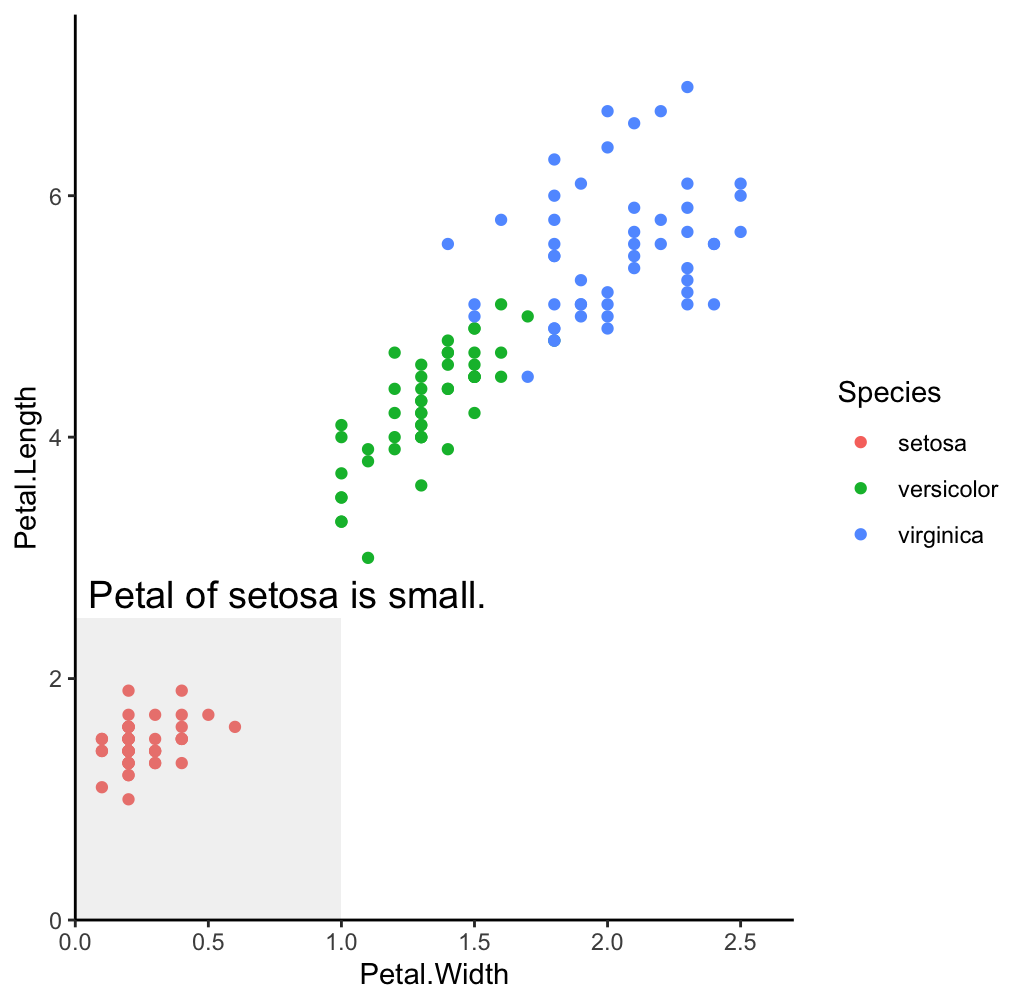
短径の追加
rectでは四角形を描画できます。
文字と合わせれば、グラフ上で注目して欲しいところを表すのに使えそうです。
p +
geom_point() +
scale_x_continuous(expand = c(0, 0), limits = c(0, 2.7)) +
scale_y_continuous(expand = c(0, 0), limits = c(0, 7.5)) +
annotate("text", x = 0.8, y = 2.7, label = "Petal of setosa is small.", color = "black", size = 5)+
annotate("rect", xmin = 0, xmax = 1, ymin = 0, ymax = 2.5, fill = "gray", alpha = 0.2)+
theme_classic()
終わりに
文字や直線、短径を追加することで、グラフの解釈がしやすくなります。
今まで論文を書く際にはイラストレータなどの他のソフトウェアで注釈を追加していましたが、Rで一気にできるのであれば作業の自動化が進みそうです。
グラフのデータ点に注釈をつける際にはまた別の方法がありますので、そちらはそちらで紹介します。
- Sepalはがく、Petalは花びら (正確な言葉遣いではないです) をさしています。↩