はじめに
大学が変わったのと年度が変わったことで、統計ソフトとグラフ作成用のソフトを使えなくなった。
これを機に統計とグラフ作成を両方Rにしようと思う。
t testのやり方もよく分かっていないので、Helpを見ながら勉強してみた。
2標本のt検定
Rでのt testはt.test()を使えば良い。
例えば1~10の数列と7~20までの数列でt testしてみる。
t.test(c(1:10), y=c(7:20))
Welch Two Sample t-test
data: c(1:10) and c(7:20)
t = -5.4349, df = 21.982, p-value = 1.855e-05
alternative hypothesis: true difference in means is not equal to 0
95 percent confidence interval:
-11.052802 -4.947198
sample estimates:
mean of x mean of y
5.5 13.5
出力にもあるとおり、デフォルトでwelch (等分散性を仮定しなくても使えるt test)になっているようだ。
あとはお馴染みの結果が返ってきている。
このようにt.test() に2群のベクタを渡せばt検定できるみたい。
簡単でよろしい。
df (自由度) が小数になっているのはwelchだから。
試しに、サンプルデータでもやってみる。
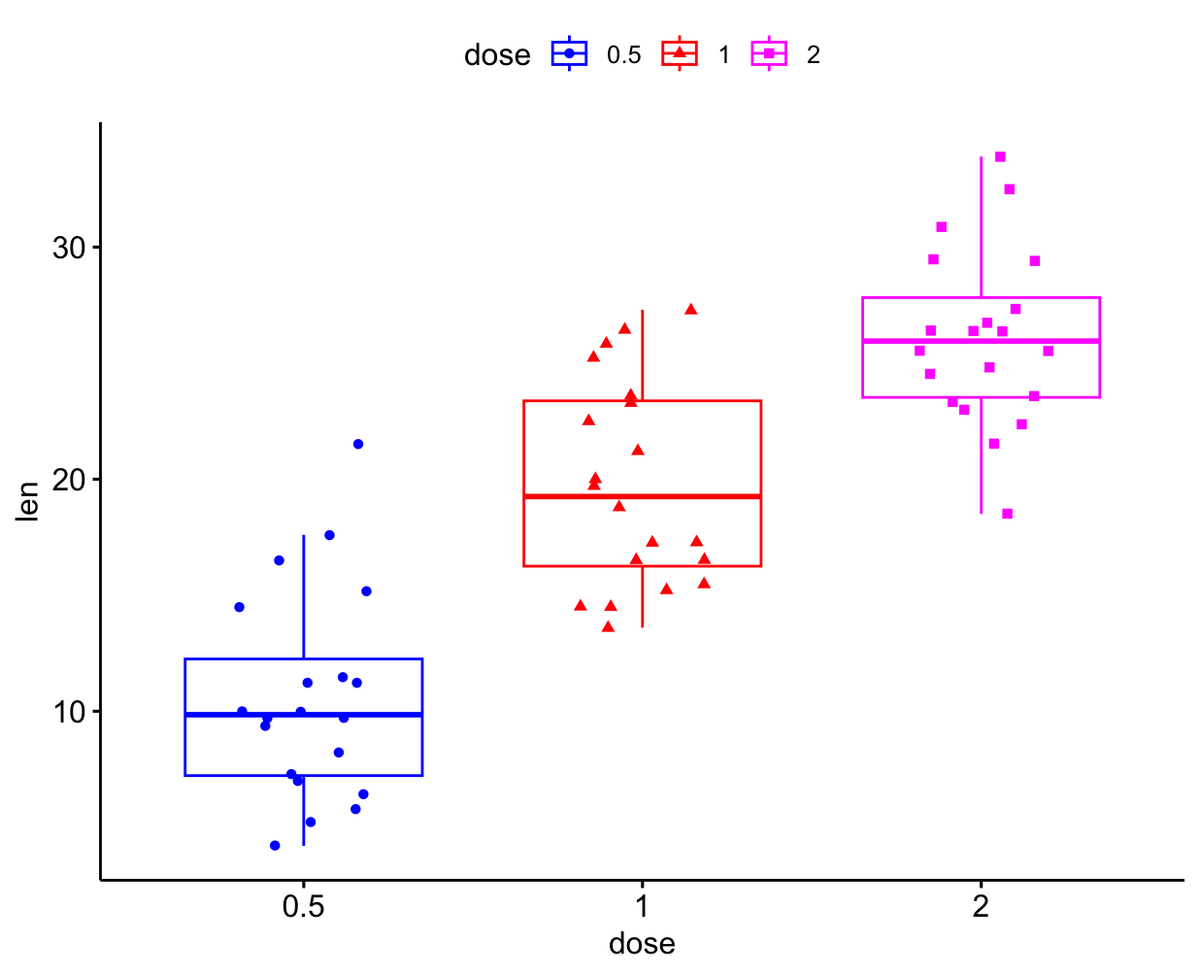
以下は前にどこかで使ったsleepデータ
勉強中のggplotで図示してみる。
testData <- sleep ggplot(data = testData, aes(x=group, y = extra))+ geom_boxplot()+ theme_classic()

前も
もう一つの書き方 fomula形式
t検定の時に、ベクタを渡す形式以外に、式で書くこともできる。 Fomula形式と呼ばれている。
従属変数 ~ 独立変数
というような形で記載することが可能であり、この際にはデータの指定が必要。
t.test(extra ~ group, data = testData)
Welch Two Sample t-test
data: extra by group
t = -1.8608, df = 17.776, p-value = 0.07939
alternative hypothesis: true difference in means between group 1 and group 2 is not equal to 0
95 percent confidence interval: -3.3654832 0.2054832
sample estimates:
mean in group 1 mean in group 2
0.75 2.33
このFomula形式で書くものといえば線形回帰が思いつく
res <- lm(extra ~ group, data = testData) summary(res)
Call:
lm(formula = extra ~ group, data = testData)
Residuals:
Min 1Q Median 3Q Max
-2.430 -1.305 -0.580 1.455 3.170
Coefficients:
Estimate Std. Error t value Pr(>|t|)
(Intercept) 0.7500 0.6004 1.249 0.2276
group2 1.5800 0.8491 1.861 0.0792 .
---
Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
Residual standard error: 1.899 on 18 degrees of freedom
Multiple R-squared: 0.1613, Adjusted R-squared: 0.1147
F-statistic: 3.463 on 1 and 18 DF, p-value: 0.07919
結果のGroup2のPrはStudentのt検定の結果と等しいはず。
t検定, ANOVA、線形回帰には本質的な違いはない (というか全て一般線形モデルの範疇内の) はずなのでこれで良い。
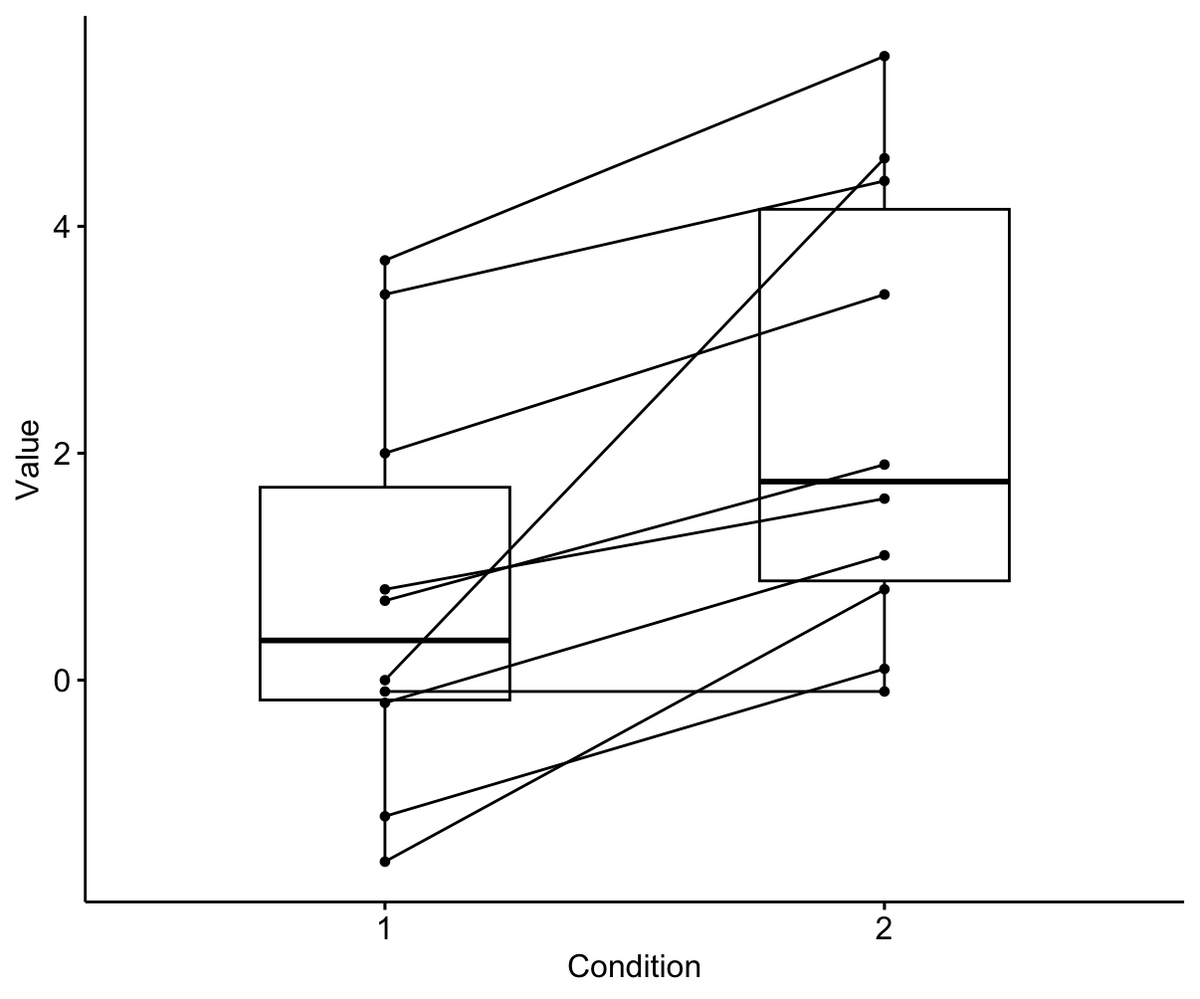
対応のあるt検定
次に対応のあるt検定をやってみる。
元のformatでは少しやりにくいが、練習と思ってやってみる。
dataの中からGourpが1, 2であるものをfilterで取り出して、格納し、対応のあるt検定
group1 <- testData %>% filter(group == "1") group2 <- testData %>% filter(group == "2") t.test(group1$extra, group2$extra, paired = T)
Paired t-test
data: group1$extra and group2$extra
t = -4.0621, df = 9, p-value = 0.002833
alternative hypothesis: true mean difference is not equal to 0
95 percent confidence interval:
-2.4598858 -0.7001142
sample estimates:
mean difference
-1.58
対応のあるt検定は差の1標本のt検定なので以下のように書いても良いはずで、実際に一致している。
t.test(group1$extra - group2$extra)
One Sample t-test
data: group1$extra - group2$extra
t = -4.0621, df = 9, p-value = 0.002833
alternative hypothesis: true mean is not equal to 0
95 percent confidence interval:
-2.4598858 -0.7001142
sample estimates:
mean of x
-1.58
感想
これなら簡単に使えそう。
one way ANOVAとか他の検定も簡単に使えそうなので、使いそうな検定を勉強していこうと思う。